Genetik und Epigenetik des Silver-Russell-Syndroms und verwandter Imprintingerkrankungen
Unter Imprintingerkrankungen werden angeborene Erkrankungen zusammengefasst, die durch gleichartige Pathomechanismen charakterisiert sind (zur Übersicht: Soellner et al., 2016). Diese umfassen verschiedene molekulare Veränderungen, die elterlich geprägte Gene und chromosomale Regionen betreffen (Abb. 1). Während die Uniparentalen Disomien (UPDs), Duplikationen/Deletionen (Copy number variations/CNVs) und Punktmutationen genomische Veränderungen darstellen, die zur gestörten Expression der betroffenen geprägten Gene führen, betreffen Epimutationen nicht die DNA-Sequenz selber, sondern die Methylierung differentiell methylierter Regionen (DMRs). Die Ursache für Epimutationen sind großteils unklar, in Einzelfällen wurden genomische Ursachen in cis oder trans (z.Bsp. benachbarte Deletionen) nachgewiesen.
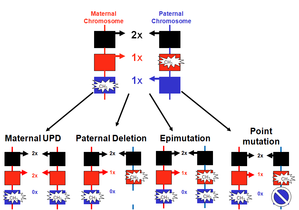
Abb. 1: Die vier molekularen Veränderungen, die bei Imprintingerkrankungen beobachtet werden.
Die genannten (Epi)Mutationstypen sind bei den meisten der derzeit bekannten 13 Imprintingerkrankungen (Abb. 2) bereits nachgewiesen, treten aber in unterschiedlichen Häufigkeiten auf (zur Übersicht: Soellner et al., 2016).
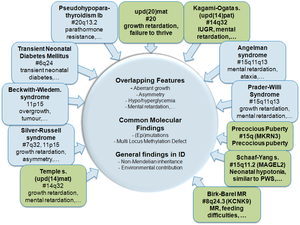
Abb. 2: Übersicht über die derzeit bekannten angeborenen Imprintingerkrankungen (blau: Erkrankungen, die vor 2013 beschrieben waren; grün: seit 2013 identifizierte Erkrankungen)
Klinisch zeigen Imprintingerkrankungen ähnliche, oft überlappende Symptome, die i.d.R. Wachstum, Asymmetrien, Stoffwechsel und Verhalten/Entwicklung betreffen. Auch weitere Ähnlichkeiten wie klinisch-diskordante monozygote Zwillinge und überlappende Multilocus-Methylierungsstörungen (MultiLocus Imprinting Disturbances) werden berichtet, für mehrere Erkrankungen wird eine Häufung durch assistierte reproduktionsmedizinische Maßnahmen diskutiert. Die meisten Fälle mit Imprintingerkrankungen treten sporadisch auf, in einzelnen Entitäten bzw. für einzelne molekulare Subtypen ist eine Familiariät berichtet (z.B. upd(15) bei Prader-Willi-/Angelman-Syndrom; Punktmutationen i.a.; CNVs). Dabei sind – in Abhängigkeit von der elterlichen Herkunft der Veränderung – Wiederholungswahrscheinlichkeiten von bis zu 50% anzugeben (Abb. 3)
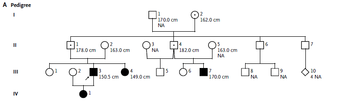
Abb. 3: Stammbaum einer Familie mit IGF2-Punktmutation (aus Begemann et al., 2015).
Schwerpunkt der Arbeitsgruppe ist das Silver-Russell-Syndrom (SRS, OMIM180860) und klinisch sowie molekular überlappende Erkrankungen (z.B. Beckwith-Wiedemann-Syndrom/BWS, Temple-Syndrom (TS)). Das SRS ist im Wesentlichen charakterisiert durch eine ausgeprägte intrauterine und postnatale Wachstumsstörung, weiterhin ist es gekennzeichnet durch kraniofaziale Auffälligkeiten wie einen normalen Kopfumfang trotz Kleinwuchses (relative Makrozephalie), prominente Stirn, dreieckförmiges Gesicht, Mikrognathie und herabgezogene Mundwinkel (zur Übersicht: Wakeling et al., 2017). Als weitere Leitsymptome gelten Asymmetrie des Körpers und Trinkschwäche. Viele Kinder zeigen Gedeihstörungen und daraus resultierende motorische Entwicklungsverzögerungen; die Nahrungsaufnahme in den ersten Lebensjahren ist oft schwierig. Die Ausprägung der o.g. Symptome ist variabel, die Diagnose ist zusätzlich durch die Abschwächung der Symptomatik mit zunehmendem Alter erschwert. Aus diesem Grunde wurde jüngst der Netchine-Harbison-Score zur klinischen Diagnostik vorgeschlagen (Wakeling et al., 2017).
Die Ätiologie des SRS ist heterogen, aber mittlerweile konnten zwei wesentliche (epi)genetische Störungen nachgewiesen werden: in ~10% der SRS-Fälle lässt sich eine maternale UPD des Chromosoms 7 (upd(7)mat)) nachweisen, ~40% zeigen eine Untermethylierung der IGF2/H19: TSS DMR (differentiell methylierte Region) auf Chromosom 11p15. Damit ist das SRS das erste humane Imprintingsyndrom, bei dem Defekte wahrscheinlich Imprinting-Regionen auf zwei verschiedenen Chromosomen betreffen. Weiterhin zeigt es molekulare bzw. klinische Überlappungen mit weiteren angeborenen Imprintingerkrankungen, wie dem Beckwith-Wiedemann-Syndrom (gegensätzliche (Epi)Mutationen in 11p15) und insbesondere dem Temple-Syndrom (Chromosom 14).
Welche Gene oder Genregionen auf Chromosom 7 an der Entstehung des SRS beteiligt sind ist derzeit trotz einer großen Zahl von Studien zu verschiedenen Chromosom 7-codierten Faktoren unklar. Nichtsdestotrotz wird die wesentliche Rolle von Chromosom 7 für das SRS auch durch mehrere chromosomale Duplikationen im kurzen Arm dieses Chromosoms unterstützt (Carrera et al., 2016).
Auch die funktionelle Relevanz der IGF2/H19: TSS DMR-Untermethylierung in 11p15 ist derzeit noch unbekannt. Dabei ist diese Veränderung, ebenso wie bei einzelnen SRS-Patienten nachgewiesene andere 11p15 Mutationen, von besonderem Interesse, da gegensätzliche Mutationstypen in 11p15 bei dem durch Großwuchs charakterisierten Beckwith-Wiedemann-Syndrom auftreten. Jüngste Daten belegen eine wesentliche Rolle des in 11p15 lokalisierten und die IGF2/H19: TSS-DMR regulierten IGF2-Gens (Begemann et al. 2015).
Klinisch weisen die Patienten mit 11p15-Epimutation den typischen SRS-Phänotyp auf, während die UPD(7)mat-Gruppe eine eher milde Symptomatik aufweist. Allerdings ist der Übergang fließend und in beiden genetischen Subgruppen sind sehr leichte und sehr schwere SRS-Phänotypen berichtet. Eine genetische Testung sollte daher immer beide (Epi)mutationstypen erfassen. Bei unauffälligem Befund sollte aufgrund der gelegentlich beobachteten Chromosomenstörungen, die dann für die weiterführende Genidentifikation hilfreich sein können, eine molekulare Karyotypisierung angeschlossen werden. In wieweit diese verschiedenen genetischen Veränderungen zukünftig unterschiedliche Therapiestrategien ermöglichen, bleibt abzuwarten, ist aber gut vorstellbar. In jedem Falle sollte – in Abhängigkeit von der klinischen Symptomatik – ein umfassender diagnostischer Algorithmus verfolgt werden, der zum einen aus Standardverfahren (zur Testung der Veränderungen auf den Chromosomen 7, 11 und 14 sowie zum Nachweis (submikroskopischer) CNVs) besteht, zum anderen Spezialanalysen umfasst (Abb. 4).

Abb. 4: Molekulardiagnostischer Algorithmus für das SRS (Eggermann et al., 2016).
Die Aktivitäten der Arbeitsgruppe verfolgen folgende Ziele:
- Aufklärung des Mutationsspektrums bei Patienten mit SRS bzw. SRS-Merkmalen und weiteren Imprintingerkrankungen.
- Welchen Anteil haben geringgradige Mosaike in anderen Geweben?
- Wie häufig sind submikroskopische Veränderungen bei SRS-Patienten?
- Verbesserung der diagnostischen Algorithmen.
- Entwicklung neuer Verfahren (Multilocus-Tests, NGS-basierte Verfahren).
- Aufklärung der Pathomechanismen, insbesondere in Hinblick auf Chromosom 7-kodierte Faktoren.
- Worin besteht der funktionelle Zusammenhang zwischen den Chromosom 7 und Chromosom 11-Veränderungen beim SRS?
- Worin liegt die Ursache für die ICR1-Hypomethylierung bei SRS-Patienten?
- Spielen funktionelle Kandidatengene eine Rolle für einzelne Patienten?
- Welche molekularen gemeinsamen Ursachen sind für Imprintingerkrankungen verantwortlich?
- Gibt es eine insbesondere prognostisch und therapeutisch relevante Genotyp-Phänotyp-Korrelation?
- Wie ist der Verlauf der SRS im Erwachsenenalter?
Diese Fragestellungen werden in Kooperation mit Kooperationspartnern im bundesweiten BMBF-geförderten Verbundprojekt "Diseases caused by imprinting defects" bearbeitet. Weiterhin leitet die Arbeitsgruppe das COST-geförderte (BM1208) "European Network of Congenital Imprinting Disorders", EUCID.net (www.imprinting-disorders.eu). Es besteht eine enge Zusammenarbeit mit nationalen und internationalen Patientenselbsthifegruppen (BKMF e.V.; Global Alliance of Silver-Russell syndrome).
Ansprechpartner:
Forschung/Diagnostik:
Prof. Dr. rer. nat. Thomas Eggermann
Tel.: 0241 80-88008
teggermannukaachende
Dr. rer. nat. Matthias Begemann
Tel.: 0241 80-80036
mbegemannukaachende
Schwerpunkt Klinische Genetik:
Dr. med. Miriam Elbracht
Tel.: 0241 80-88013
mielbrachtukaachende
Schwerpunkt Methodenentwicklung:
Dr. rer. nat. Florian Kraft
Tel.: 0241 80-80036
fkraftukaachende
Schwerpunkt Qualitätssicherung/EMQN:
Dr. rer. nat. Katja Eggermann
Tel.: 0241 80-88008
keggermannukaachende
Literatur:
Begemann M, Zirn B, Santen G, Wirthgen E, Soellner L, Büttel HM, Schweizer R, van Workum W, Binder G, Eggermann T. Paternally Inherited IGF2 Mutation and Growth Restriction. N Engl J Med. 2015 Jul 23;373(4):349-56
Carrera IA, de Zaldívar MS, Martín R, Begemann M, Soellner L,EggermannT. Microdeletions of the 7q32.2 imprinted region are associated with Silver-Russell syndrome features. Am J Med Genet A. 2016 Mar;170(3):743-9
Eggermann K, Bliek J, Brioude F, Algar E, Buiting K, Russo S, Tümer Z, Monk D, Moore G, Antoniadi T, Macdonald F, Netchine I, Lombardi P, Soellner L, Begemann M, Prawitt D, Maher ER, Mannens M, Riccio A, Weksberg R, Lapunzina P, Grønskov K, Mackay DJ, Eggermann T. EMQN best practice guidelines for the molecular genetic testing and reporting of chromosome 11p15 imprinting disorders: Silver-Russell and Beckwith-Wiedemann syndrome. Eur J Hum Genet. 2016 Oct;24(10):1377-87.
Soellner L, Begemann M, Mackay DJ, Grønskov K, Tümer Z, Maher ER, Temple IK, Monk D, Riccio A, Linglart A, Netchine I, Eggermann T. Recent Advances in Imprinting Disorders. Clin Genet. 2017 Jan;91(1):3-13
Wakeling EL, Brioude F, Lokulo-Sodipe O, O'Connell SM, Salem J, Bliek J, Canton AP, Chrzanowska KH, Davies JH, Dias RP, Dubern B, Elbracht M, Giabicani E, Grimberg A, Grønskov K, Hokken-Koelega AC, Jorge AA, Kagami M, Linglart A, Maghnie M, Mohnike K, Monk D, Moore GE, Murray PG, Ogata T, Petit IO, Russo S, Said E, Toumba M, Tümer Z, Binder G, Eggermann T, Harbison MD, Temple IK, Mackay DJ, Netchine I. Diagnosis and management of Silver-Russell syndrome: first international consensus statement. Nat Rev Endocrinol. 2017 Feb;13(2):105-124
Links:











