Forschung
Die Erbinformation ist in Eukaryonten im Zellkern vom Rest der Zelle abgetrennt. Diese räumliche Trennung ist durch die Kernhülle gegeben, die aus zwei Membranen, der innere und äußere Kernmembran, besteht. Eingebettet in dieses Zweimembransystem sind Kernporenkomplexe, die Transportöffnungen der Kernhülle. Dies sind hochmolekulare Proteinkomplexes – 125 MDa in Vertebraten – die den molekularen Austausch zwischen dem Zytoplasma und dem Zellkerninneren bewerkstelligen.
Schematische Darstellung des Zellkerns und der Kernhülle
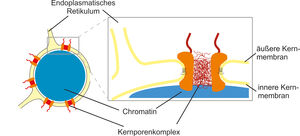
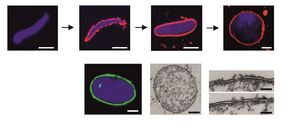
Während in Hefezellen die Kernhülle während der Zellteilung intakt bleibt, bricht in tierischen Zellen die Kernhülle mit dem Eintritt in die Mitose zusammen. Am Ende der Mitose baut sie sich um das Chromatin der Tochterzellen wieder auf. Wir untersuchen die molekulare Mechanismen des Wiederaufbaus von Kernhülle und Kernporenkomplexen sowie die Chromatindekondensierung. Mit Eiextrakten aus Xenopus laevis können wir diese Prozesse im Reagenzglas nachstellen, biochemisch manipulieren und untersuchen. So identifizieren wir Schlüsselkomponenten, definieren ihre Funktion in den Prozessen und wie sie reguliert werden.
Frösche in unseren Aquarien und die Eier, die sie legen

Kernporenaufbau und Funktion
Kernporen vermitteln den Protein-, RNA- und Metabolittransport zwischen Zytoplama und Zellkern. Mit einer Masse von 125 MDa sind sie die größten Proteinkomplexe, die wir kennen. Der schrittweise koordinierte Aufbau dieser Megastrukturen aus mehr als 500 einzelnen Proteinen und, wie sie sich in die Kernhülle integrieren, ist ein faszinierendes Beispiel für die zelluläre Selbstorganisation. Mit biochemischen und zellbiologischen Methoden definieren wir den Bauplan dieser Komplexe und ihre Funktion in den verschiedenen Transportprozessen. Wir untersuchen die Membraninteraktion von Kernporenproteinen und wie diese zum Aufbau der Kernporen beiträgt.
Obwohl Kernporen in allen Zellen eine essentielle Funktion besitzen, verursachen einige Mutationen in Kernporenproteinen sehr spezifische Pathologien, z. B. eine arterielle Fibrillation, nephrotisches Syndrom mit fatalen Nierenversagen in Kindern oder das AAA-Syndrom, bei dem die Patienten eine Achalsie, Alacrima und Morbus Addinson zeigen. Wir charakterisieren, wie spezifische Mutationen in Kernporenproteinen sehr spezifische Zelltypen betrifft, und definieren die molekularen Mechanismen der beschriebenen Pathologien.
Ausbildung einer Kernhülle und Kernporenkomplexe im Reagenzglas
Weiterführende Literatur
Vollmer B, Lorenz M, Moreno-Andres D, Bodenhöfer M, De Magistris P, Astrinidis SA, Schooley A, Flötenmeyer M, Leptihn S, and Antonin W (2015). Nup153 recruits the Nup107-160 complex to the inner nuclear membrane for interphasic nuclear pore complex assembly. Developmental Cell, 33 (6): 717-728.
Braun DA, Sadowski CE, Kohl S, Lovric S, Astrinidis SA, Pabst WL, Gee HY, Ashraf S, Lawson JA, Shril S, Airik M, Tan W, Schapiro D, Rao J, Choi WI, Hermle T, Kemper MJ, Pohl M, Ozaltin F, Konrad M, Bogdanovic R, Büscher R, Helmchen U, Serdaroglu E, Lifton RP, Antonin W, Hildebrandt F (2016). Mutations in nuclear pore genes NUP93, NUP205 and XPO5 cause steroid-resistant nephrotic syndrome. Nature Genetics, 48 (4): 457-465.
Weberruss M and Antonin W (2016). Perforating the nuclear boundary - how nuclear pore complexes assemble. Journal of Cell Science 129 (24): 4439-4447
Chromatindekondensierung am Ende der Mitose
In tierischen Zellen ist das mitotische Chromatin in Chromosomen fünfzigfach dichter gepackt als in der Interphase. Am Ende der Mitose, wenn sich ein Zellkern wieder ausbildet, muss das Chromatin wieder entpackt werden, so das es transkribiert und repliziert werden kann. Obwohl dies ein essentieller Prozess im Lebenszyklus der Zellen ist, ist über ihn wenig bekannt. Mit einem zellfreien System identifizieren und charakterisieren wir Chromatindekondensierungsfaktoren.
Chromatindekondensierung im Reagenzglas

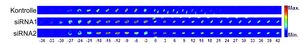
Durch life cell imaging, oft in Kombination mit RNAi basierten Screeningmethoden, identifizieren wir Schlüsselkomponenten der Chromatindekondensierung, die Dynamik der Chromatindekondensierung und wie sie mit anderen zellulären Prozessen, die zeitglich ablaufen, koordiniert ist.
Chromatindekondensierung in Zellen
Weiterführende Literatur:
Magalska, A, Schellhaus, AK, Moreno-Andres, D, Zanini, F, Schooley, A, Sachdev, R, Schwarz, H, Madlung, J, Gerken, J and Antonin, W (2014). RuvB-like ATPases function in chromatin decondensation at the end of mitosis. Developmental Cell, 31 (3): 305-318.
Schellhaus, AK, Magalska, A, Schooley, A, and Antonin, W (2015). A cell free assay to study chromatin decondensation at the end of mitosis. Journal of Visual Experiments (JoVE), 106: doi: 10.3791/53407.
Antonin W and Neumann H (2016). Chromosome condensation and decondensation during mitosis. Current Opinion in Cell Biology, 40:15-22.
Vesikelbildung in der Kernhülle durch Herpesviren
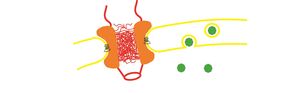
Obwohl Kernporenkomplexe die eigentlichen Transportkanäle der Kernhülle darstellen, ignorieren Herpesvieren diesen Weg. Sie verlassen den Zellkern, indem sie an der inneren Kernmembran Vesikel bilden. Diese Vesikel schnüren sich in den Zwischenraum zwischen innerer und äußerer Kernmembran ab und fusionieren mit der äußeren Kernmembran. Dabei wird das Viruspartikel im Zytosol freigesetzt. Wir untersuchen mit minimalen Membransystemen wie GUVs (giant unilammelar vesicles), welche Proteine die Vesikelbildung und -abschnürung vermitteln und wie sie das bewerkstelligen.
Herpesviren bilden Kernmembranvesikel
Weiterführende Literatur:
Lorenz M, Vollmer B, Unsay JD, Klupp BG, García-Sáez AJ, Mettenleiter TC, and Antonin W (2015). A Single Herpesvirus Protein can mediate Vesicle Formation in the Nuclear Envelope. Journal of Biological Chemistry, 290 (11): 6962-6974.
Zeev-Ben-Mordehai, T, Weberruß, M, Lorenz, M, Cheleski, J, Hellberg, T, Whittle, K, El-Omari, K, Vasishtan, D, Dent, K C, Harlos, K, Hagen, W, Klupp, BG, Antonin, W, Mettenleiter, TC, and Grünewald, K (2015). Crystal structure of the herpesvirus nuclear egress complex provides insights into inner nuclear membrane remodelling. Cell Reports, 13 (12): 2645-2652.




